Exossomos de Mastócitos na Comunicação Imune Intercelular
Exossomos derivados de mastócitos são vesículas extracelulares de 30–150 nm que transportam proteínas, lipídios e RNAs funcionais, atuando como mediadores críticos da comunicação intercelular no sistema imune. Segundo a International Society for Extracellular Vesicles (ISEV) , essas nanovesículas representam um dos mecanismos mais sofisticados de transferência de informação molecular entre células.
Um estudo publicado no Nature Cell Biology , utilizando o ZetaView da Particle Metrix, demonstrou que exossomos podem transferir mRNAs e microRNAs funcionais entre mastócitos, estabelecendo um novo paradigma de comunicação intercelular. Essa descoberta abriu caminho para investigações sobre o cargo molecular específico dessas vesículas e seu papel na amplificação de respostas imunes.
Nesse contexto, o ZetaView PMX 110 foi utilizado na caracterização física dos exossomos isolados de mastócitos humanos ( HMC-1 ), realizando Nanoparticle Tracking Analysis (NTA) para determinar distribuição de tamanho e concentração de partículas. A análise confirmou vesículas com diâmetro predominante de 50–150 nm (média ~100 nm) e estruturas arredondadas típicas, em conformidade com as diretrizes MISEV2018 para caracterização de vesículas extracelulares.
O estudo de Liang et al. (2018) identificou o receptor de quimiocinas CCR1 como hub gene em exossomos de mastócitos, revelando um potencial mecanismo de transferência horizontal de receptores que pode amplificar respostas alérgicas e inflamatórias.
Este artigo analisa os achados metodológicos e funcionais desse estudo, com foco na caracterização física por Nanoparticle Tracking Analysis (NTA), pipeline bioinformático para identificação de genes diferencialmente expressos (DEGs) e validação experimental do CCR1 como molécula central na sinalização exossomal de mastócitos.
Principais tópicos abordados neste artigo
- Metodologia de isolamento e caracterização física de exossomos via NTA (ZetaView PMX 110)
- Pipeline bioinformático: dataset GSE25330, análise de DEGs e construção de redes PPI
- Perfil transcriptômico diferencial: 2121 DEGs entre exossomos e células HMC-1
- CCR1 como hub gene no módulo de sinalização de quimiocinas
- Validação experimental por qRT-PCR e Western blot
- Implicações da costimulação FcεRI/CCR1 na degranulação mastocitária
- Hipótese de transferência horizontal de receptores via exossomos
Metodologia do Estudo Original: Isolamento e Análise de Exossomos de Mastócitos
Isolamento e Caracterização Física de Exossomos por NTA
O isolamento de exossomos da linhagem HMC-1 (mastócitos humanos) foi realizado por centrifugação diferencial, conforme protocolo estabelecido por Théry et al. (2006) . O processo envolveu centrifugações sequenciais a 300×g, 2.000×g e 10.000×g para remoção de células mortas e debris, seguidas de ultracentrifugação a 100.000×g por 70 minutos para pelletização dos exossomos. Adicionalmente, mastócitos derivados de medula óssea murina (BMMCs) foram cultivados para validação experimental por qRT-PCR.
A caracterização física foi realizada por Nanoparticle Tracking Analysis (NTA) utilizando o sistema ZetaView PMX 110 (Particle Metrix, Meerbusch, Alemanha), equipado com software de captura de vídeo e rastreamento de partículas. A análise revelou distribuição de tamanho predominante na faixa de 50–150 nm, consistente com o perfil esperado para exossomos.
A técnica de NTA permite quantificação precisa de concentração e distribuição de tamanho de nanopartículas em suspensão, sendo considerada padrão-ouro para caracterização de vesículas extracelulares segundo as diretrizes MISEV2018 da ISEV.
A morfologia foi confirmada por microscopia eletrônica de transmissão (TEM), evidenciando estruturas vesiculares arredondadas de 30–150 nm. A expressão dos marcadores exossomais TSG101 e CD63 foi validada por Western blot, confirmando a identidade e pureza das preparações.
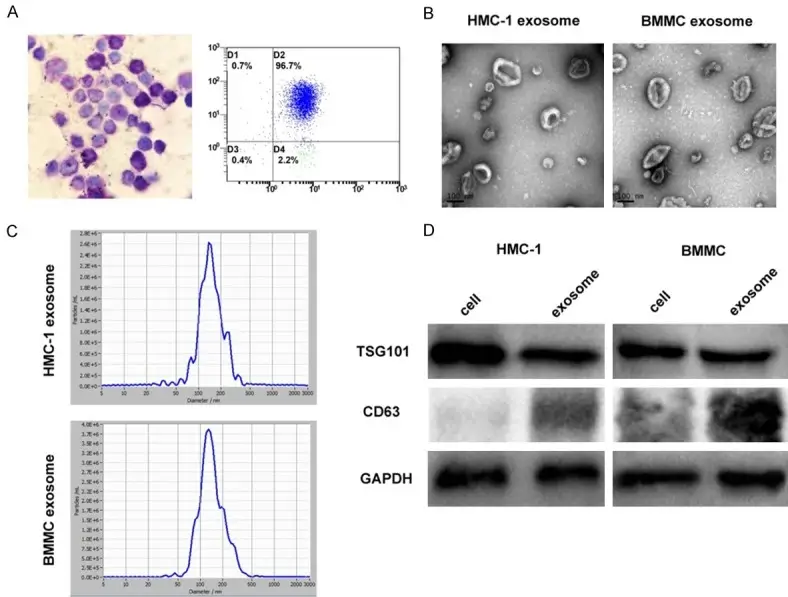
[Figura 1: Identificação e caracterização de exossomos derivados de mastócitos. A. Mastócitos derivados de medula óssea (BMMCs) com grânulos púrpura-avermelhados por coloração com azul de toluidina (400×); expressão de CD117 e FcεRI detectada por citometria de fluxo. B. Micrografias de transmissão eletrônica dos exossomos isolados revelando estruturas arredondadas com diâmetro de 30-150 nm. C. Distribuição de tamanho dos exossomos medida por Nanoparticle Tracking Analysis (NTA). D. Western blot mostrando expressão dos marcadores exossomais TSG101 e CD63.]
Pipeline Bioinformático para Identificação de Genes Diferencialmente Expressos
O estudo utilizou o dataset GSE25320 da plataforma Gene Expression Omnibus (GEO), baseado no array Affymetrix Human Genome U133 Plus 2.0. O dataset compreendeu 4 amostras de RNA exossomal HMC-1 e 4 amostras de RNA celular HMC-1.
O pré-processamento dos dados foi realizado com pacotes Bioconductor em R, incluindo correção de background e normalização. A análise diferencial utilizou o pacote limma, com critérios de significância estatística definidos como |log2 fold change| >2 e P ajustado <0.01.
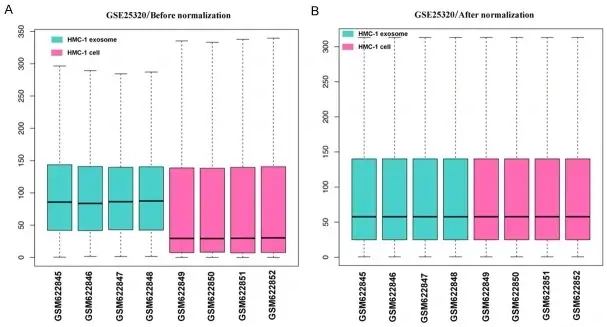
[Figura 2: Box plot dos dados de expressão antes e após normalização. O eixo horizontal representa os nomes das amostras e o eixo vertical representa os níveis de expressão. A linha preta em cada caixa representa os níveis de expressão de cada amostra. (A) Dados antes da normalização e (B) dados após normalização.]
Para anotação funcional, foram empregadas análises de Gene Ontology (GO) e Kyoto Encyclopedia of Genes and Genomes (KEGG) via plataforma DAVID. A construção de redes de interação proteína-proteína (PPI) utilizou o banco STRING com scores de interação >0.95 (todos os DEGs) e >0.7 (genes up-regulados), visualizadas no software Cytoscape. A identificação de módulos funcionais foi realizada com o plugin Cluster ONE.
Validação Experimental: qRT-PCR e Western Blot
Sete genes foram selecionados para validação experimental: CCR1 , CD9 , KIT , TGFBR1 , TLR9 , TPSAB1 e TPSB2 . A seleção incluiu genes com relevância funcional previamente estabelecida (CCR1 na sinalização de quimiocinas), marcadores específicos de mastócitos (KIT, TPSAB1, TPSB2), marcador exossomal (CD9) e genes aleatoriamente selecionados entre up e down-regulados (TLR9, TGFBR1).
A expressão foi quantificada por qRT-PCR com normalização para GAPDH pelo método 2 -ΔΔCt , tanto em HMC-1 quanto em BMMCs. A validação proteica de CCR1 foi realizada por Western blot especificamente em lisados de células HMC-1 e seus exossomos derivados.
Resultados: Perfil Transcriptômico Diferencial de Exossomos de Mastócitos
Genes Diferencialmente Expressos e Enriquecimento Funcional
A análise identificou 2.121 genes diferencialmente expressos entre exossomos HMC-1 e células parentais, sendo 843 up-regulados e 1.278 down-regulados nos exossomos. Essa assimetria sugere um mecanismo seletivo de empacotamento de transcritos nas vesículas.
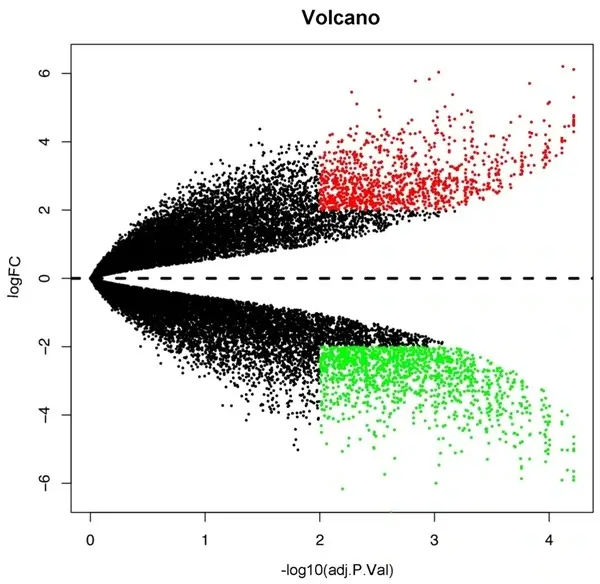
[Figura 3: Volcano plot dos genes diferencialmente expressos em exossomos e células HMC-1. Vermelho: genes significativamente up-regulados; verde: genes significativamente down-regulados. FC, fold change.]
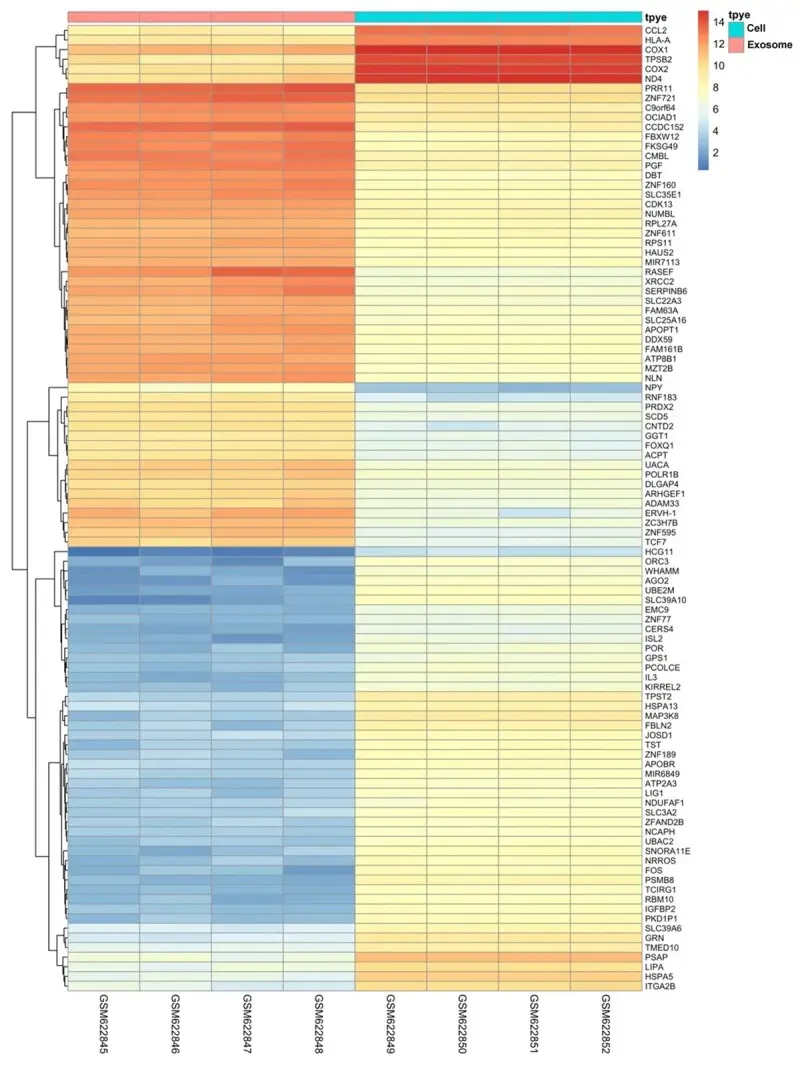
[Figura 4: Heatmap dos 100 genes diferencialmente expressos mais significativos, gerado por análise de clustering hierárquico.]
Os genes up-regulados nos exossomos apresentaram enriquecimento significativo nas seguintes categorias GO-Biological Process:
- Fibrinólise (GO:0042730)
- Transmissão sináptica química (GO:0007268)
- Metabolismo de xenobióticos (GO:0006805)
- Adesão celular (GO:0007155)
- Via de sinalização mediada por citocinas (GO:0019221)
Para os genes down-regulados , o enriquecimento predominou em processos intracelulares como sumoilação proteica, replicação de DNA, coesão de cromátides-irmãs e processamento de antígenos via MHC classe I.
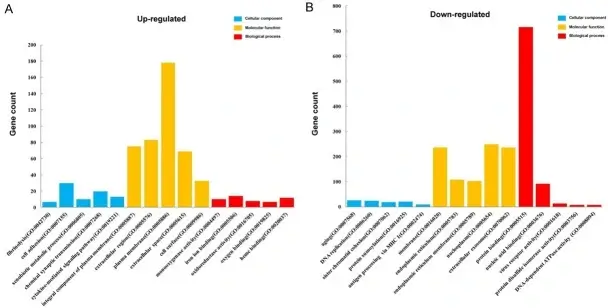
[Figura 5: Análise de Gene Ontology e termos GO significativamente enriquecidos. (A) DEGs up-regulados e (B) DEGs down-regulados em exossomos HMC-1 versus células HMC-1.]
O enriquecimento de genes relacionados a sinalização de citocinas e resposta imune nos exossomos sugere um papel ativo dessas vesículas na modulação de respostas inflamatórias e alérgicas.
A análise de vias KEGG revelou enriquecimento dos genes up-regulados em circadian entrainment ( hsa04713 ), carcinogênese química ( hsa05204 ), moléculas de adesão celular ( hsa04514 ) e interação neuroativo ligante-receptor ( hsa04080 ). Os down-regulados concentraram-se em lisossomo ( hsa04142 ), processamento proteico no retículo endoplasmático ( hsa04141 ) e ciclo celular ( hsa04110 ).
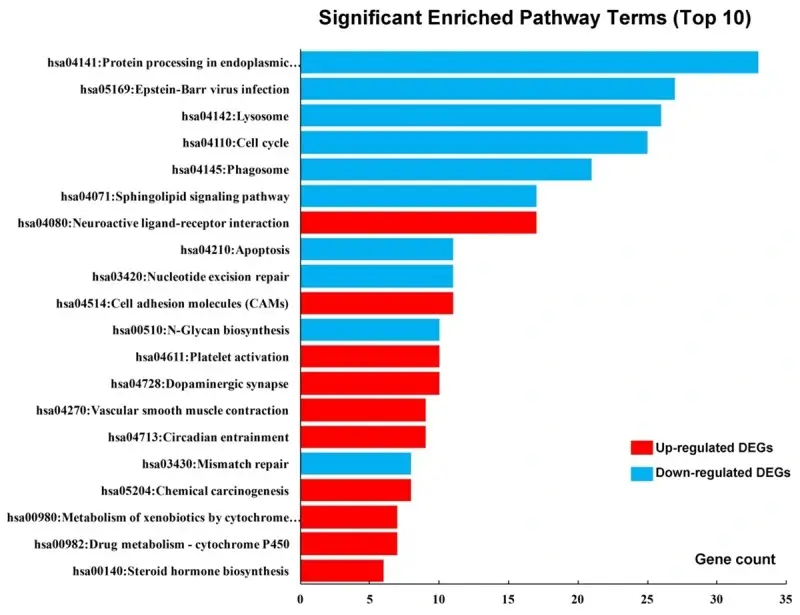
[Figura 6: Termos de vias significativamente enriquecidos dos DEGs. Enriquecimento funcional e de vias de sinalização dos DEGs realizado por análises KEGG Pathway.]
Rede PPI e Identificação de CCR1 como Hub Gene
A rede PPI construída com todos os DEGs (score >0.95) compreendeu 243 nós e 395 interações, com predominância de genes down-regulados. Uma segunda rede focada exclusivamente nos genes up-regulados (score >0.7) revelou 138 nós e 320 arestas.
Os dez genes com maior grau de conectividade na rede up-regulada foram:
| Gene | Grau |
|---|---|
| ADCY2 | 23 |
| CXCL10 | 15 |
| GNAT3 | 15 |
| S1PR4 | 13 |
| CCL28 | 13 |
| TAS2R4 | 12 |
| OXER1 | 12 |
| P2RY4 | 12 |
| CCR1 | 12 |
| CORT | 12 |
| Módulo | Termo | Descrição | P-value |
|---|---|---|---|
| 1 | hsa04062 | Via de sinalização de quimiocinas | 2.00×10⁻³ |
| 1 | GO:0006955 | Resposta imune | 2.90×10⁻³ |
| 1 | GO:0006935 | Quimiotaxia | 3.29×10⁻³ |
| 2 | GO:0016567 | Ubiquitinação proteica | 6.30×10⁻¹⁰ |
| 2 | GO:0035556 | Transdução de sinal intracelular | 4.47×10⁻⁴ |
A análise modular identificou dois clusters significativos. O Módulo 1 , contendo CCR1, apresentou enriquecimento robusto para via de sinalização de quimiocinas, resposta imune e quimiotaxia. O Módulo 2 associou-se a ubiquitinação proteica e transdução de sinal intracelular.
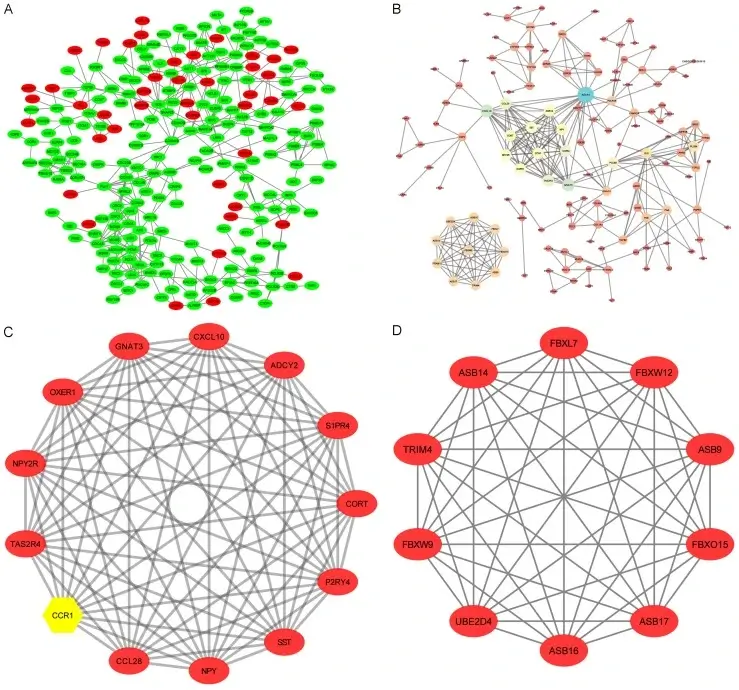
[Figura 7: Rede de interação proteína-proteína (PPI) e análise modular dos DEGs. A. Nós vermelhos representam DEGs up-regulados e nós verdes representam genes down-regulados. B. Rede PPI construída para todos os genes up-regulados com score PPI >0.7. C, D. Dois módulos obtidos da rede PPI dos DEGs up-regulados; CCR1 (amarelo) identificado como hub gene.]
Validação Experimental de CCR1 e Outros Marcadores
A validação por qRT-PCR confirmou a expressão diferencial dos sete genes selecionados, com concordância entre os resultados bioinformáticos e experimentais tanto em mastócitos humanos (HMC-1) quanto murinos (BMMCs). A análise por Western blot, realizada especificamente em HMC-1, confirmou níveis proteicos de CCR1 significativamente elevados nos exossomos em comparação às células parentais.
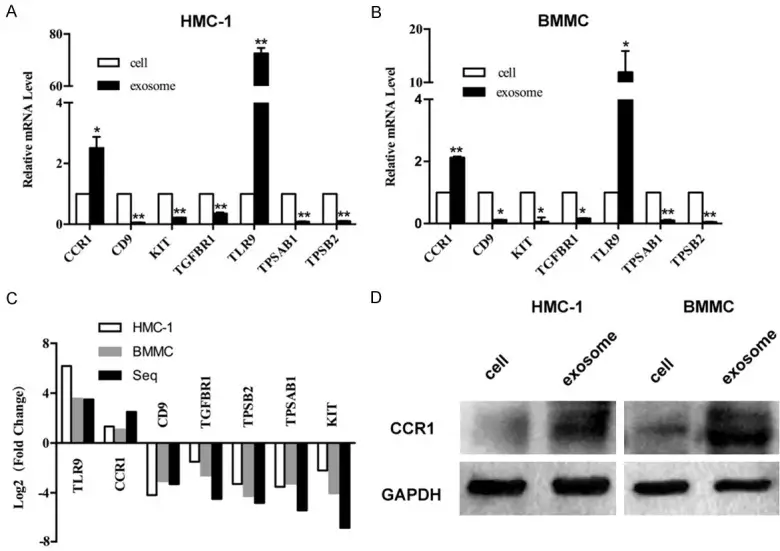
[Figura 8: Validação dos DEGs em exossomos de mastócitos. A e B. qRT-PCR quantitativo para verificar fold changes dos DEGs em exossomos derivados de mastócitos versus mastócitos, *P<0.05, **P<0.01. C. Fold-changes dos RNAs de mastócitos versus RNAs exossomais verificados por qRT-PCR, confirmando alta confiabilidade dos genes candidatos identificados. D. Western blot confirmando níveis de expressão proteica de CCR1 em mastócitos e exossomos; CCR1 elevado nos exossomos derivados de mastócitos. GAPDH usado como controle interno.]
Genes up-regulados nos exossomos (confirmados):
- CCR1 – receptor de quimiocinas CC
- TLR9 – receptor toll-like 9
Genes down-regulados nos exossomos (confirmados):
- CD9 – tetraspanina (marcador exossomal clássico)
- KIT – receptor de fator de células-tronco (marcador de mastócitos)
- TGFBR1 – receptor de TGF-β tipo I
- TPSAB1 e TPSB2 – triptases (marcadores específicos de mastócitos)
A down-regulação de CD9 nos exossomos, apesar de ser considerado marcador exossomal, sugere heterogeneidade na composição vesicular e reforça a importância de múltiplos marcadores na caracterização.
Discussão: Implicações do CCR1 Exossomal na Sinalização Imune
CCR1 e Costimulação FcεRI na Degranulação de Mastócitos
O CCR1 é um receptor de quimiocinas CC que reconhece ligantes como CCL3 (MIP-1α), CCL5 (RANTES) e CCL7 (MCP-3). Estudos anteriores demonstraram que mastócitos das vias aéreas de pacientes asmáticos expressam CCR1 e CCR4 em níveis elevados, conforme documentado por Amin et al. (2005) .
A relevância funcional do CCR1 em mastócitos foi estabelecida por experimentos de costimulação. Aye et al. (2012) demonstraram que a ativação simultânea de FcεRI (receptor de alta afinidade para IgE) e CCR1 resulta em degranulação mastocitária significativamente maior do que a estimulação de qualquer receptor isoladamente.
Essa costimulação induz secreção de TNF-α, TGF-β1 e IL-6, além de amplificar a liberação de histamina e outros mediadores pré-formados. O mecanismo envolve convergência de vias de sinalização intracelular, incluindo fosforilação de PLCγ e mobilização de cálcio.
Hipótese de Transferência Horizontal de CCR1 via Exossomos
O enriquecimento de CCR1 em exossomos de mastócitos levanta uma hipótese mecanística importante: essas vesículas podem transferir receptores funcionais para mastócitos vizinhos ou outros tipos celulares imunes, amplificando a sensibilidade local a quimiocinas.
Se confirmada, essa transferência horizontal representaria um mecanismo de "priming" exossomal, onde mastócitos em estado basal receberiam CCR1 adicional via exossomos de células ativadas, tornando-se mais responsivos a estímulos subsequentes. Isso poderia explicar, em parte, a hiperreatividade observada em condições alérgicas crônicas.
A transferência de receptores funcionais via exossomos já foi documentada para KIT em adenocarcinoma pulmonar, conforme demonstrado pelo próprio grupo de pesquisa em estudo anterior. Essa evidência reforça a plausibilidade biológica da hipótese de transferência de CCR1.
Do ponto de vista patológico, exossomos CCR1-ricos poderiam contribuir para:
- Amplificação de respostas alérgicas mediadas por IgE
- Perpetuação de inflamação crônica em asma e rinite
- Recrutamento aumentado de células efetoras via sinalização de quimiocinas
- Polarização de respostas imunes para perfil Th2
Limitações do Estudo e Perspectivas Futuras
O estudo apresenta limitações que devem ser consideradas na interpretação dos resultados. O uso da linhagem celular HMC-1, derivada de leucemia mastocitária, pode não refletir completamente a biologia de mastócitos primários teciduais. A validação em BMMCs murinos fortalece os achados, mas diferenças espécie-específicas não podem ser descartadas.
A demonstração funcional da transferência de CCR1 via exossomos permanece como lacuna experimental crítica. Estudos futuros deverão avaliar uptake de exossomos CCR1-positivos por células receptoras, persistência do receptor transferido na membrana, funcionalidade (binding de ligantes, ativação de vias downstream) e efeito na degranulação de mastócitos naive.
Do ponto de vista terapêutico, antagonistas de CCR1 como BX471 poderiam ser investigados como moduladores da sinalização exossomal em contextos alérgicos. Alternativamente, estratégias de depleção de exossomos ou bloqueio de seu uptake representam abordagens potenciais para controle de inflamação mastócito-dependente.
Conclusão: CCR1 como Alvo na Comunicação Exossomal de Mastócitos
A caracterização transcriptômica de exossomos de mastócitos, combinando análise por NTA (ZetaView) e pipeline bioinformático robusto, identificou CCR1 como hub gene no módulo de sinalização de quimiocinas. A validação experimental por qRT-PCR e Western blot confirmou o enriquecimento desse receptor nas vesículas em comparação às células parentais.
Esses achados posicionam o CCR1 exossomal como potencial mediador de amplificação de respostas imunes via comunicação intercelular. A hipótese de transferência horizontal de receptores abre perspectivas para investigação de biomarcadores e alvos terapêuticos em doenças alérgicas e inflamatórias.
A integração de técnicas de caracterização física de alta resolução, bioinformática de larga escala e validação experimental representa um modelo metodológico aplicável ao estudo de vesículas extracelulares em outros contextos imunológicos e patológicos.
Perguntas Frequentes sobre Exossomos de Mastócitos e CCR1
O que são exossomos derivados de mastócitos?
São vesículas extracelulares de 30–150 nm liberadas por mastócitos, contendo proteínas, lipídios, mRNAs e microRNAs. Atuam como mediadores de comunicação intercelular, podendo transferir informação molecular para células receptoras e modular respostas imunes.
Qual a função do CCR1 em mastócitos?
CCR1 é um receptor de quimiocinas CC (reconhece CCL3, CCL5, CCL7) expresso na superfície de mastócitos. Sua ativação, especialmente em costimulação com FcεRI, amplifica a degranulação e liberação de mediadores inflamatórios como histamina, TNF-α e IL-6.
Como os exossomos de mastócitos foram caracterizados no estudo?
Por Nanoparticle Tracking Analysis (NTA) com sistema ZetaView PMX 110, microscopia eletrônica de transmissão (TEM) e Western blot para marcadores TSG101 e CD63. A análise molecular utilizou microarray (Affymetrix U133 Plus 2.0) e validação por qRT-PCR.
O que significa CCR1 ser um hub gene?
Na análise de redes de interação proteína-proteína (PPI), hub genes são nós com alto grau de conectividade, indicando centralidade funcional. CCR1 apresentou grau 12 no módulo de sinalização de quimiocinas, sugerindo papel coordenador nessa via.
Qual a relevância clínica do enriquecimento de CCR1 em exossomos?
Se exossomos podem transferir CCR1 funcional para outras células, isso representaria um mecanismo de amplificação de respostas alérgicas. Mastócitos receptores teriam maior sensibilidade a quimiocinas, potencialmente contribuindo para hiperreatividade em asma e outras condições atópicas.
Quais são as limitações do estudo de Liang et al. (2018)?
Uso de linhagem celular HMC-1 em vez de mastócitos primários, ausência de demonstração funcional de transferência de CCR1 via exossomos, e necessidade de validação in vivo dos mecanismos propostos.
Existem aplicações terapêuticas potenciais desses achados?
Antagonistas de CCR1 (ex.: BX471) poderiam modular a sinalização exossomal em doenças alérgicas. Adicionalmente, CCR1 exossomal poderia servir como biomarcador de ativação mastocitária em fluidos biológicos.
Estudo original: https://pmc.ncbi.nlm.nih.gov/articles/PMC5835801/
Leitura Complementar
- Caracterização de Vesículas Extracelulares por NTA – metodologias e padronização segundo MISEV2018
- Degranulação de Mastócitos – mecanismos moleculares e regulação por receptores FcεRI e GPCRs
- Sinalização de Quimiocinas na Inflamação Alérgica – papel de CCR1, CCR3 e CCR4 em asma e rinite